生命現象を知るためには、生物の体の中で多様な働きを担うタンパク質の理解が不可欠です。これまで研究に必要なタンパク質は、大腸菌や酵母などの生きた細胞を使って生産されてきましたが、試験管内で無細胞転写・翻訳できる「PUREシステム」の登場により、夾雑物や分解酵素を含まない非常にクリーンな系でタンパク質合成ができるようになりました。ELSIメンバーらからなる国際研究チームは、市販のPUREシステムを利用し、反応条件の最適化及び生成物の純度を高めた 高効率ディスプレイ法の開発に成功しました。
(Graphical abstract adopted from Reyes et al., Biotechnol Bioeng, 118(4):1736-1749. (2021))
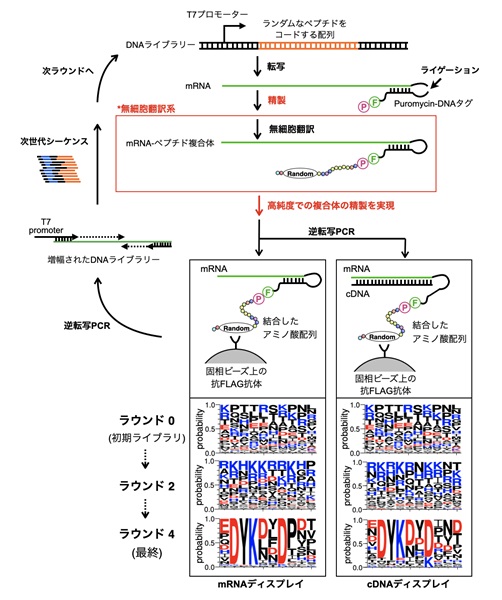
無細胞転写・翻訳システムは、反応溶液の条件が柔軟で、生きた細胞と比べて制約が少ないことから、試験管内でのタンパク質発現に不可欠なツールとなっています。特に従来の細胞抽出液ではなく、精製した翻訳関連因子のみで再構成された「PUREシステム」は、夾雑物や分解酵素を含まないため、安定したRNAやタンパク質の合成を可能にしました。グラスゴー大学工学系研究科のSabrina Galiñanes Reyes大学院生(研究当時。現ELSI技術支援員)及びELSIの藤島皓介准教授らをはじめとする国際研究チームは、市販のPUREシステムをmRNAディスプレイ法に適用し、1年以上かけて反応条件の最適化及び生成物の純度を高めるための2つのゲル精製ステップを検討しました。これらの改良により、化学的に結合したmRNA(遺伝子型)とそれがコードするタンパク質(表現型)の複合体形成が大幅に向上しました。さらに、mRNA領域の二次構造形成を防いで、非特異的相互作用を最小限に抑えるためにcDNA鎖を逆転写で合成するcDNAディスプレイ法も検討しました。
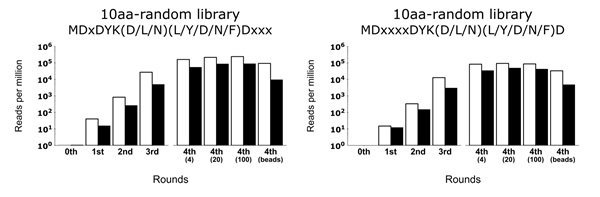
これらのディスプレイ法は、1回の実験で膨大な数のタンパク質分子(最大で1013個)をスクリーニングすることが可能です。今回は2種類のディスプレイ法の性能を比較評価するために市販の抗FLAG抗体を対象としました。これは、既知の短いFLAG結合モチーフ(DYKxxD)の特徴を利用したものです。研究チームは、約1.7×1012個のランダムな10アミノ酸長のペプチド配列から始めて合計4回のセレクションを行いました(図1)。

抗FLAG抗体に対するセレクションの各ラウンドで濃縮した上位の配列を次世代シーケンサーにより明らかにすることで、最終的にmRNA/cDNAディスプレイのいずれの方法でもFLAG結合モチーフが濃縮することがわかりました。面白いことにモチーフはランダム配列中の異なる2箇所(2番目の残基と5番目の残基を先頭とする)でそれぞれ濃縮していることがわかり、いずれの場合でもリード数上位50配列の大半を占めるには、最低3回セレクションを回せば十分であることがわかりました(図2)。これは過去のディスプレイ法と比べても非常に効率が良いことを示唆しています。また2種類のディスプレイ法を比較すると、それぞれ十分な濃縮が確認できましたが、mRNAディスプレイの方がわずかに濃縮効率が高いことが明らかになりました(図3)。
一方、ゲル切り出し精製の工程を省いた対照実験では、mRNAとタンパク質の複合体形成が不十分で、目的とは異なる長さの配列も増加していました。このことから精製プロセスを加えてタンパク質ライブラリの高い純度を保つことで、正しいアプタマー(*)配列を迅速に濃縮できることがわかりました。一方、他のRNAやタンパク質など雑多な因子が混ざっている状況下では、cDNAディスプレイの方がmRNAディスプレイよりも有用であることもわかりました。これは、mRNA領域がcDNAでマスキングされ、非特異的な相互作用を最小限に抑えられているからだと予想されます。
本手法は多様なポリペプチド鎖の機能と配列空間を探るための強力なツールとなります。また、本手法を利用することで、例えば、生命の起源研究に関連する原始的なタンパク質の機能空間の理解や、将来の生体分子ツールやペプチド医薬品開発のための新規タンパク質アプタマーの探索など、生物学に関連した幅広いニーズに答えることが可能です。
*アプタマー:特定の標的分子に特異的に結合する核酸分子やペプチド。今回の場合は抗FLAG抗体に結合するペプチド配列を指す)
| 掲載誌 | Biotechnology and Bioengineering |
| 論文タイトル | PURE mRNA display and cDNA display provide rapid detection of core epitope motif via high‐throughput sequencing |
| 著者 | Sabrina Galiñanes Reyes1,2,3, Yutetsu Kuruma1,2,4, Mai Fujimi1, Masako Yamazaki5, Sumie Eto1,5, Shota Nishikawa1,6, Satoshi Tamaki5, Asaki Kobayashi7, Ryo Mizuuchi4,8, Lynn Rothschild9, Mark Ditzler9, Kosuke Fujishima1,10 |
| 所属 | 1. Earth‐Life Science Institute, Tokyo Institute of Technology, Meguro‐ku, Tokyo, Japan 2. Extra‐cutting‐edge Science and Technology Avant‐garde Research Program, Japan Agency for Marine‐Earth Science and Technology, Kanagawa, Japan 3. James Watt School of Engineering, The University of Glasgow, Glasgow, UK 4. JST, PRESTO, Saitama, Japan 5. MOLCURE Inc., Shinagawa, Tokyo, Japan 6. School of Life Science and Technology, Tokyo Institute of Technology, Tokyo, Japan 7. SABNP, Univ Evry, INSERM U1204, Université Paris‐Saclay, Evry, France 8. Komaba Institute for Science, The University of Tokyo, Meguro‐ku, Tokyo, Japan 9. Center for the Emergence of Life, NASA Ames Research Center, Moffett Field, California, USA 10. Graduate School of Media and Governance, Keio University, Fujisawa, Japan |
| DOI | 10.1002/bit.27696 |
| 出版日 | 2021年1月27日 |
