地球の生命が他の惑星でも生存可能かどうかを知ることは、生命の起源を理解するためだけではなく、他の惑星をテラフォーム(地球化)する可能性を考える上でも必要です。東京工業大学地球生命研究所 (ELSI) の研究者らからなる研究グループは、生物学の大規模なデータベースと計算科学のツールを組み合わせて、さまざまな惑星環境で必要な化学物質を生成する生命の能力を予測するフレームワークを作成しました。本研究の成果は、生物と惑星環境の相互作用をより定量的に予測することを可能にし、宇宙生物学に不可欠な基盤を提供します。
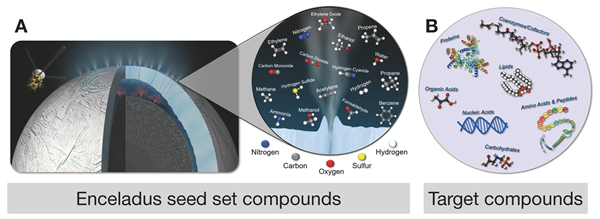
地球の生物が他の惑星で生存可能かどうかを調べるために、研究者たちは実験室で他の惑星の条件を再現したり、地球上で他の惑星と類似した地域を訪れたりして実験を行います。これらのアプローチはどちらも貴重な知見をもたらしますが、費用面でも時間の面でも多大なコストがかかります。もし、お金をかけて旅行やフィールドワークをし、時間をかけて生物の成長を観察する代わりに、生物学者が日常的に収集して大規模なデータベースにアップロードしているようなデータや、惑星探査機ですでに収集されているようなデータに基づいて、他の惑星における地球生命体の生存率を予測する別の方法があったとしたらどうでしょうか。東京工業大学地球生命研究所 (ELSI) のHarrison Smith研究員(研究当時・アリゾナ州立大学School of Earth and Space Exploration研究員)は、アリゾナ州立大学の研究者とともに、生化学と惑星観測のデータを統合して、他の惑星における地球の生命の生存率を予測するためのフレームワークを開発しました。また、そのフレームワークを土星の第2衛星エンケラドゥスにおいて実行し、生物の生存可能性を計算できることも実証しました。
研究グループは、Joint Genome Institute (JGI) のデータを使用し、エンケラドゥスの海と同じpH範囲で生息する地球の生物のサブセットを特定しました。次に、JGIデータを介してこれらの生物の触媒能力を特定しました。この情報とエンケラドゥスのさまざまな観測データから同定された化学物質を組み合わせることで、各生物が触媒できるすべての反応を特定することができました。さらに、ネットワーク拡張プロセスを使用してシミュレーションを実行し、それを新しい化学物質を生成できなくなるまで繰り返し、生成された化学物質を、系統樹上で生物の生存のために広く必要とされる化学物質のリストと比較しました。生物がリスト内のすべての化学物質を作ることができない場合、それは生存不能であると見なしました。
エンケラドゥスの環境についてわかっている知見をふまえ、エンケラドゥスの海と同じpH範囲で生息する地球生物にこの手法を適用すれば、これらの生物がエンケラドゥスで生存可能かどうかを調べることができます。研究グループが実際に解析したところ、残念ながら、生存可能ではありませんでした。おそらく、生命を構成する重要な要素の1つであるリンがないことが理由です。リンは、特に低レベルでは検出が難しいため、実際にはエンケラドゥスにある程度の量は存在していると考えても不合理ではありませんが、リンを含めてシミュレーションを行っても、生物が生成する化学物質は生存に必要な量に達しませんでした。
次に研究グループは、ネットワーク拡張技術の変形を使用して、これらの生物が生存能力を獲得するためには、エンケラドゥスでどのような化合物が検出される必要があるのかを調べました。その結果、少なくともさらに数種類の化合物が必要であり、それらの化合物は、現在エンケラドゥスに存在が知られているものよりも大きく、より複雑でなければならないことがわかりました。しかし、すべての希望が失われたわけではありません。現在エンケラドゥスで観測されている化合物は、機器の感度によって制限されていますが、これは今後のミッションで改善される可能性があるからです。
研究グループが作成したフレームワークは、あらゆる惑星環境や生物反応のネットワークに適用できます。データ収集が向上するにつれて、より精度の高い予測を行うことができ、生命とその起源である惑星の相互依存関係についての洞察がさらに広がることが期待されます。
| 掲載誌 | Astrobiology |
| 論文タイトル | Seeding Biochemistry on Other Worlds: Enceladus as a Case Study |
| 著者 | Harrison B. Smith1,*, Alexa Drew1, John F. Malloy1, and Sara Imari Walker1–4 |
| 所属 | 1 School of Earth and Space Exploration, Arizona State University, Tempe, Arizona, USA. 2 ASU-SFI Center for Biosocial Complex Systems, Arizona State University, Tempe, Arizona, USA. 3 Beyond Center for Fundamental Concepts in Science, Arizona State University, Tempe, Arizona, USA. 4 Santa Fe Institute, Santa Fe, New Mexico, USA. *Current affiliation: Earth-Life Science Institute, Tokyo Institute of Technology, Tokyo, Japan |
| DOI | 10.1089/ast.2019.2197 |
| 出版日 | 2021年2月4日 |
