生物は、タンパク質を構成するために20種類のアミノ酸(便宜上、標準アミノ酸と呼ぶ)を利用します。標準アミノ酸は、かつてはもっと少なかったのですが、生物の進化とともに、最終的に20種類に「標準化」した可能性があります。東京工業大学地球生命研究所(ELSI)に集まり研究を実施した国際的かつ学際的研究チームは、この標準アミノ酸に着目し、適応特性が時間とともにどのように進化するのかをモデル化しました。その結果、ごく少数でも標準アミノ酸を含むセットは、それ以外の膨大な種類のアミノ酸よりも物理化学的特性が優れていることを突き止めました。つまり、どれだけ多様なアミノ酸の組み合わせからスタートしても、最終的には地球の生命が利用しているような標準アミノ酸の組み合わせにたどり着く可能性があることを初めて示しました。
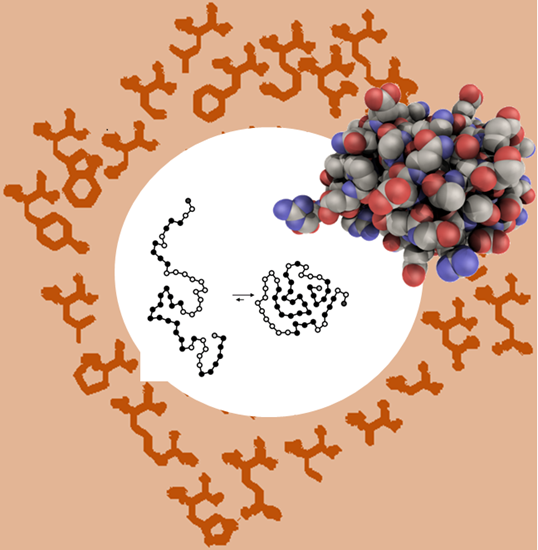
Melissa Ilardo博士(現在ユタ大学に所属)らによる今回の研究では、生物において最も重要な構成要素の一つであるタンパク質を構成するアミノ酸に着目し、アミノ酸の組み合わせが進化とともにどう変わるかをモデル化しました。そして、どのようなアミノ酸の組み合わせからスタートしても、現在の20種類の標準アミノ酸に帰結することを示しました。
バクテリアから人間まで、地球上の全ての生命は20種類のコード化された標準アミノ酸を使ってタンパク質を合成しています。この標準アミノ酸の組み合わせは、おそらく最初は、もっと限られた少数の組み合わせからスタートし、進化の過程で徐々に拡張されてきたはずです。研究チームは、この組み合わせがどのように拡張してきたのかを調べました。
地球や宇宙で発見される可能性のあるアミノ酸の数は数百万種類に及びますが、それぞれが特有の化学的特性を持っています。研究チームの先行研究では、標準アミノ酸のセットとランダムに選択したアミノ酸のセットを比較し、10億とおりの組み合わせの中で、標準アミノ酸の化学的特性の多様性を上回る組み合わせはたった1つだけであることを示しました。
そこで今回研究チームは、進化の初期段階における少数のアミノ酸の組み合わせを分析しました。合計1,913種類の標準アミノ酸を含む”ありえた”アミノ酸のライブラリを使用し、3~20種類の範囲での組み合わせを試しました。20種類のアミノ酸の組み合わせは1048通りありますが、これは宇宙全体の星の数の約1024個を大幅に上回る数です。そこで統計的手法を用い、さらに、標準アミノ酸の物理化学的性質の組み合わせの適応度を基準として、数十億個のその他の組み合わせの適応度と比較しました。Jim Cleaves博士は「天然に存在するアミノ酸の数を考慮すると、この天文学的な数の組みあわせ問題を解く唯一の方法は計算科学によるアプローチしかないと考えた」と述べています。
その結果、生命が利用する標準アミノ酸は、機能性のタンパク質を生み出すために必要な化学的性質に基づいて進化の過程で選択されてきた可能性が高いことがわかりました。
実際に、標準アミノ酸を1つまたは少数しか含まない仮想的なアミノ酸の組み合わせでも、際立った適応性があることを発見しました。逆に膨大な数の組み合わせの中から、標準アミノ酸のセットに匹敵する化学的性質の多様性を持つ組み合わせを見つけることは困難でした。したがって今回の結果は、進化の過程で標準アミノ酸が遺伝子にコードされるごとに、他のアミノ酸では示すことができない適応度を獲得してきたことを示します。そして、最終的に現在の地球上の生命に見られる標準アミノ酸の組み合わせに収束していくことを示唆しています。
本モデルはアミノ酸自身が持つ物理化学的特性に基づいています。したがって、仮に地球外生命体が存在した場合、アミノ酸を基に機能性のタンパク質を合成しているとすれば、地球上の生命が利用する標準アミノ酸の組み合わせに収斂している可能性があることを意味しています。本論文の共著者であるマックス・プランク分子細胞生物学・遺伝学研究所のRudrarup Bose氏は、「生命は単なる偶然の事象の結果ではないのかもしれない。むしろ、生命の進化の方向性を司る普遍的な法則が存在するのかもしれない」と、述べています。
| 掲載誌 | Scientific Reports |
| 論文タイトル | Adaptive Properties of the Genetically Encoded Amino Acid Alphabet Are Inherited from Its Subsets |
| 著者 | Melissa Ilardo1, Rudrarup Bose2, Markus Meringer3, Bakhtiyor Rasulev4, Natalie Grefenstette5, James Stephenson6,7, Stephen Freeland8, Richard J. Gillams9,10, Christopher J. Butch9,11,12 & H. James Cleaves II 9,12,13* |
| 所属 | 1. University of Utah Hematology, UC Berkeley Integrative Biology, George and Dolores Eccles Institute of Human Genetics, 15 N 2030 E, Room: 3240, Salt Lake City, UT, 84112, USA. 2. National Institute of Science Education and Research Bhubaneswar, P.O. Jatni, Khurda, 752050, Odisha, India. 3. German Aerospace Center (DLR), EarthObservation Center (EOC), Münchner Straße 20, 82234, Oberpfaffenhofen-Wessling, Germany. 4. Department of Coatings and Polymeric Materials, North Dakota State University, Fargo, ND, 58108, USA. 5. Department of Chemistry, University College London, 20 Gordon Street, London, WC1H 0AJ, UK. 6. European Molecular Biology Laboratory–European Bioinformatics Institute, Wellcome Trust Genome Campus, Hinxton, UK. 7. Wellcome Trust Sanger Institute, Wellcome Trust Genome Campus, Hinxton, UK. 8. University of Maryland, Baltimore County, 1000 Hilltop Circle, Baltimore, MD, 21250, USA. 9. Earth-Life Science Institute, Tokyo Institute of Technology, 2-12-1-IE-1 Ookayama, Meguro-ku, Tokyo, 152-8550, Japan. 10. Structural Genomics Consortium, Nuffield Department of Medicine, University of Oxford, Old Road Campus Research Building, Oxford, OX3 7DQ, UK. 11. Department of Chemistry, Emory University, 1515 Dickey Drive, Atlanta, GA, USA. 12. Blue Marble Space Institute for Science, 1001 4th Ave, Suite 3201, Seattle, WA, 98154, USA. 13. Institute of Advanced Study, 1 Einstein Drive, Princeton, NJ, 08540, USA. |
| DOI | 10.1038/s41598-019-47574-x |
| 出版日 | 2019年8月28日 |
